La présente étude donne suite à la présentation de Jérome Waldispuhl, professeur au Department of Computer Science — McGill University, portant sur l’utilisation du crowdsourcing pour résoudre des problèmes de génomique. Elle vise à donner une perspective globale du crowdsourcing appliqué à la bioinformatique par une description des différents systèmes et une revue d’application concrètes. En conclusion, des cas concrets montrent que le crowdsourcing peut être une solution viable pour certains problèmes de bioinformatique.
Le 12 février dernier, Jérome Waldispuhl, professeur au Department of Computer Science — McGill University, nous présentait les réalisations de son groupe de recherche visant à utiliser le jeu en ligne comme moyen pour solutionner certains problèmes en génomique, une approche qui relève du « crowdsourcing ». L’objectif de la présente étude est de donner une perspective plus globale sur l’utilisation du crowdsourcing pour résoudre des problèmes de bioinformatique. Dans un premier temps, le crowdsourcing sera définit et une description des différents systèmes utilisés sera donnée. En second lieu, une revue d’exemples concrets de problèmes bioinformatiques qui ont été soumis au crowsourcing sera effectuée, avec une évaluation de l’efficacité du procédé utilisé et du succès obtenu.
Dans le contexte de la bioinformatique, le crowdsourcing
peut se définir comme l’acte de prendre une tâche normalement
accomplie par un expert et de la faire exécuter dans le cadre
d’un appel public par un grand nombre d’individus, généralement
sans expertise (Good et Su, 2013). Les
principales raisons pour faire appel au crowdsourcing
sont les suivantes :
- Limites computationnelles : certains problèmes ne peuvent être investigué à l’aide d’algorithmes de manière exhaustive dû aux nombre énorme de combinaisons possibles, menant à des solutions approximées; on fait appel à l’intervention humaine pour optimiser d’avantage ces solutions (Cooper et al., 2010; Kawrykow et al., 2012);
- Limites dans l’appropriation d’un contexte : malgré les progrès accomplies, la reconnaissances de contextes visuels et l’interprétation de textes est encore une tâche qui ne se soumet pas facilement à l’automatisation; l’humain est souvent plus efficace pour ce genre de tâche (Burger et al., 2012; Candido dos Reis et al., 2015; Cooper et al., 2010);
- Stimuler l’innovation : il s’agit d’aller au‑delà des méthodes courantes connues pour résoudre les problèmes existants; la créativité est une caractéristique propre à l’être humain (Cooper et al., 2010; Lakhani et al., 2013).
On remarque d’emblée que tous font référence aux limites de l’automatisation. Le champ de l’Intelligence Artificielle vise à simuler efficacement les habiletés humaines de manière automatique, mais certaines de ces habiletés sont toujours difficile à reproduire. Or le volume et la complexité des tâches en bioinformatique qui demandent une intervention humaine dépassent parfois la capacité de traitement des experts, d’où le recours au crowdsourcing (Good et Su, 2013).
Plusieurs systèmes de crowdsourcing existent; ils peuvent être subdivisés en deux classes principales selon le type de problème à résoudre (Good et Su, 2013) : « micro-tâches » et « méga‑tâches ».
Si un problème peut être résolu relativement sans difficulté et rapidement — en quelques secondes ou quelques minutes — à l’aide d’une courte liste d’instructions, alors ce problème peut être défini comme une micro‑tâche (Good et Su, 2013). Les micro‑tâches se prêtent bien à un grand volume de tâches qui peuvent être accomplies par la plupart des individus, et elles tendent à donner des résultats équivalents à ceux d’un expert lorsque le processus est validé par redondance et agrégation. La redondance implique d’avoir plusieurs exemplaires de la même tâche qui seront résolus par des individus différents; l’agrégation est la comptabilisation des diverses réponses pour chacun des problèmes. Cinq types de systèmes s’appliquent aux micro‑tâches : volontariat, jeu occasionnel, marché de micro‑tâches, travail forcé et système éducationnel (Good et Su, 2013).
Le volontariat — ou « Citizen
Science » — consiste tout simplement à faire appel à des
volontaires pour résoudre les micro‑tâches. La motivation de
base est l’altruisme, i.e. la satisfaction de participer
à une cause d’intérêt ou d’importance. L’exemple classique est
le recensement des oiseaux, où un groupe de citoyens volontaires
compte le nombre et le type d’oiseaux dans un secteur donné (Good et Su, 2013; Kawrykow
et al., 2012); cette approche permet de couvrir un
grand territoire rapidement et le fait d’avoir plusieurs
observateurs constitue la redondance. Des tâches qui se prêtent
bien au volontariat mentionnons l’annotation d’images et
l’annotation de textes.
La motivation pour le jeu est le divertissement. Or il y a un intérêt pour tenter de rendre productives les innombrables heures « futiles » passées à jouer par la communauté importante des individus s’adonnant aux jeux (Cooper et al., 2010; Kawrykow et al., 2012). Le type de jeu qui s’applique aux micro‑tâches est le jeu simple, ne demandant pas d’investissement à long terme, où chaque « partie » est une micro‑tâche qui se complète relativement rapidement. Dans ce type de jeu, les actions sont typiquement hautement répétitives et sont utilisées pour migrer d’état en état vers une solution. Il y a également nécessité de préserver l’aspect divertissant en faisant plus ou moins abstraction du domaine d’origine du problème. Ce type de crowdsourcing requiert un investissement initial dans la conception et le développement du jeu. De plus, une méthode objective est souvent requise pour valider les résultats (Kawrykow et al., 2012).
Dans ce système, il s’agit d’offrir sur un marché des micro‑tâches à résoudre en échange d’une rémunération à l’unité. La motivation est le gain en argent, et en conséquence l’offre et la demande pour les micro‑tâches évoluera en fonction de la difficulté de la tâche vs la rémunération offerte. Le marché de micro‑tâches le plus notoire est Amazon Mechanical Turk (AMT); il est à noter que les tâches sur cette plateforme se doivent d’être formulées très simplement, sous la forme de choix de réponses (Burger et al., 2012; Good et Su, 2013). L’annotation d’images ou de textes est une tâche de choix pour les marchés de micro‑tâches.
Comme le nom l’indique, il s’agit d’imposer l’accomplissement d’une micro‑tâche en l’imbriquant dans un flux de travail déjà existant d’une autre tâche. Ce type de système requiert donc d’avoir le plein contrôle sur un tel flux de travail. L’exemple typique est l’association de mots et d’images avec ReCAPTCHA, qui s’insère dans le processus d’authentification sécurisée d’un site web (Good et Su, 2013).
Avec ce système de crowdsourcing, on a recours pour accomplir les micro‑tâches à des experts, mais des experts « en devenir »; les tâches font partie du curriculum et constituent à la fois une pratique de l’apprentissage et la réalisation d’un travail concret et utile (Good et Su, 2013). La communauté cible est évidemment plus restreinte, se limitant aux étudiants dans le domaine concerné dans certaines institutions.
Lorsque la tâche à accomplir est complexe ou d’un haut niveau de difficulté, et qu’elle demanderais même d’un expert des semaines ou des mois de travail, alors la tâche peut être qualifiée de méga‑tâche (Good et Su, 2013). Pour l’accomplissement de ce type de tâche, le crowdsourcing sert à recruter un petit nombre d’individus talentueux parmi une large population de candidats. On mise ici sur les différences de perspectives, de talents et de compétences au sein de la communauté pour trouver des solutions originale à des problèmes difficiles. Deux types de systèmes sont pertinents pour les méga‑tâches : le jeu investi et le concours d’innovation (Good et Su, 2013).
Dans ce type de jeu, la méga‑tâche est souvent proposée comme un « casse‑tête » ou « puzzle ». Le joueur a accès à un environnement interactif riche permettant l’exploration du problème sur le long terme. L’apprentissage des règles du jeux — i.e. des procédures pour obtenir une solution — peut potentiellement demander beaucoup de temps, sans compter les niveaux d’entraînement préalables avant de pouvoir contribuer à la résolution de véritables problèmes. Le développement de ce type de jeu est souvent complexe et demande un investissement de ressources considérables. Enfin, l’évaluation des résultats nécessite une fonction objective automatique pour l’obtention d’un score (Cooper et al., 2010; Good et Su, 2013).
Le concours d’innovation propose la méga‑tâche comme un défi au grand public, invitant la soumission de solutions. Les solutions sont revues selon des critères biens définis, et les meilleures solutions sont récompensées par des prix en argent. Ce type de crowdsourcing nécessite des ressources suffisantes pour offrir des récompenses intéressantes; par contre, il permet de mobiliser rapidement beaucoup d’heures de recherche et développement par rapport à l’investissement (Good et Su, 2013).
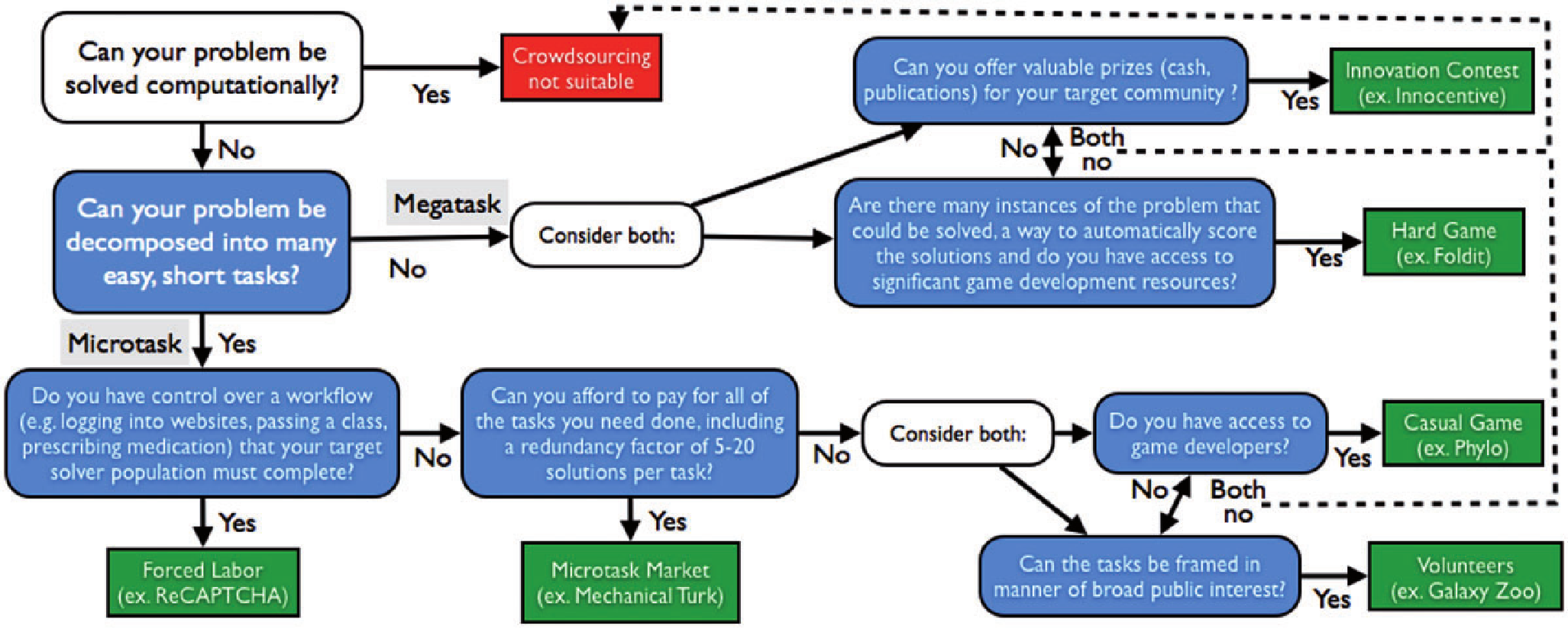
La Table 1 résume les caractéristiques de chacun des systèmes de crowdsourcing. La Figure 1 propose un processus de décision pour la sélection d’un système approprié pour un problème donné. Le point essentiel à considérer est la difficulté de parvenir à une solution computationnelle. De plus, les résultats de la méthode de crowdsourcing utilisée se doivent d’être comparable ou supérieurs aux méthodes heuristiques existantes (Kawrykow et al., 2012).
| Classe
de tâche |
Type |
Conditions |
Motivation |
Contrôle
de qualité |
| Micro |
Volontariat |
Tâche d’intérêt pour le
public, grand volume de tâches |
Altruisme |
Redondance et agrégation
(R&A) |
| Micro |
Jeu
occasionnel |
Accès à des développeurs
de jeux, grand volume de tâches |
Divertissement |
R&A, fonction de score |
| Micro | Marché de
micro‑tâches |
Accès à des fonds
suffisants pour le volume de travail |
Argent |
R&A |
| Micro | Travail forcé |
Contrôle d’un flux de
travail que la communauté cible a besoin d’exécuter |
Complétion d’une autre
tâche importante |
R&A |
| Micro | Système
éducationnel |
Double objectif
d’éducation et d’accomplissement de tâche |
Éducation |
R&A |
| Méga |
Jeu investi |
Accès à des développeurs
de jeux, problème avec une fonction objective d’évaluation |
Divertissement | Fonction de score |
| Méga | Concours d’innovation | Accès à des ressources
suffisantes pour offrir une récompense intéressante |
Argent | Revue par les créateurs du
concours |
Cette section présente des exemples concrets de problèmes bioinformatiques qui ont été soumis au crowdsourcing, soit un exemple par type de système.
L’application Cell Slider (https://www.cellslider.net/) — en opération sur la plateforme de crowdsourcing Zooniverse de 2012 à 2014 — avait pour mission de faire annoter par des volontaires des échantillons de tissues de patients atteints du cancer et ayant été traités en essai clinique. La classification d’images de populations de cellules requiert l’œil entraîné d’un expert, ce qui impose une limite quant au traitement d’un volume important d’échantillons, d’où le recours au crowdsourcing (Candido dos Reis et al., 2015). Pour chaque série d’images associée à un échantillon, on soumet aux bénévoles quatre types de tâche de classification : identification (Figure 2) et quantification du nombre de cellules cancéreuses, quantification de la proportion entre cellules saines et cellules cancéreuses, et classification de l’intensité de la coloration des cellules marquées positives. Dans chaque cas, on présente à l’utilisateur une liste de choix de réponses pré-définies accompagnées d’exemples visuels. Le contrôle de qualité est effectué par redondance et agrégation.
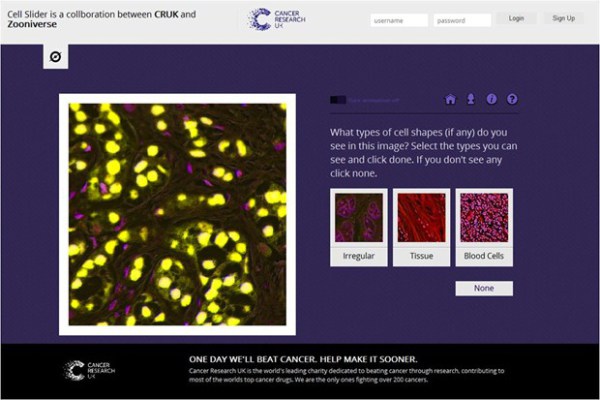
Au cours de ses deux années d’opération, Cell Slider à reçu la visite de 98,293 volontaires qui ont classifié 180,172 images dérivées de 12,326 échantillons de tissue (Candido dos Reis et al., 2015). Les résultats montrent que les tumeurs évaluées par les non‑experts sont associées à la survie du patient de manière similaire aux évaluations faites par des pathologistes entraînés; le crowdsourcing est donc une solution viable pour ce type de tâche.
Le jeu en ligne Phylo (https://phylo.cs.mcgill.ca/) — développé par l’équipe du professeur Jérome Waldispuhl — vise à améliorer des alignements locaux multiples de séquences. Les alignements déterminés automatiquement ne sont pas toujours optimaux dû à des limites computationnelles, ce qui peut avoir un impact pour certaines applications (Kawrykow et al., 2012). Le procédé consiste à sélectionner un ensemble d’alignements dont le degré de confiance est en‑deçà d’un certain seuil, puis de soumettre les séquences correspondantes à un alignement interactif manuel effectué par le joueur. Le jeu s’apparente à Tetris, les nucléotides étant représentées par des blocs de couleurs, repoussant la complexité scientifique à l’arrière plan pour mettre de l’avant le côté abstrait et divertissant (Figure 3). Un arbre phylogénétique simple donne quand même une idée du contexte scientifique. Le joueur est présenté avec un score initial d’alignement qu’il doit tenter de battre dans un temps limite. Le joueur passe de tableau en tableau, le niveau de difficulté croissant avec le nombre de séquences présentées. Les meilleurs joueurs sont récompensés par une mention publique dans un « Hall of Fame ». Le jeu inclus également un aspect « role‑playing » par la possibilité de sélectionné le type de maladie auquel les alignements appartiennent. Les alignements proposés proviennent de la base de données UCSC et porte sur 44 espèces différentes de vertébrés. Les alignements améliorés sont mis à jour dans la base de données d’origine après agrégation et évaluation.
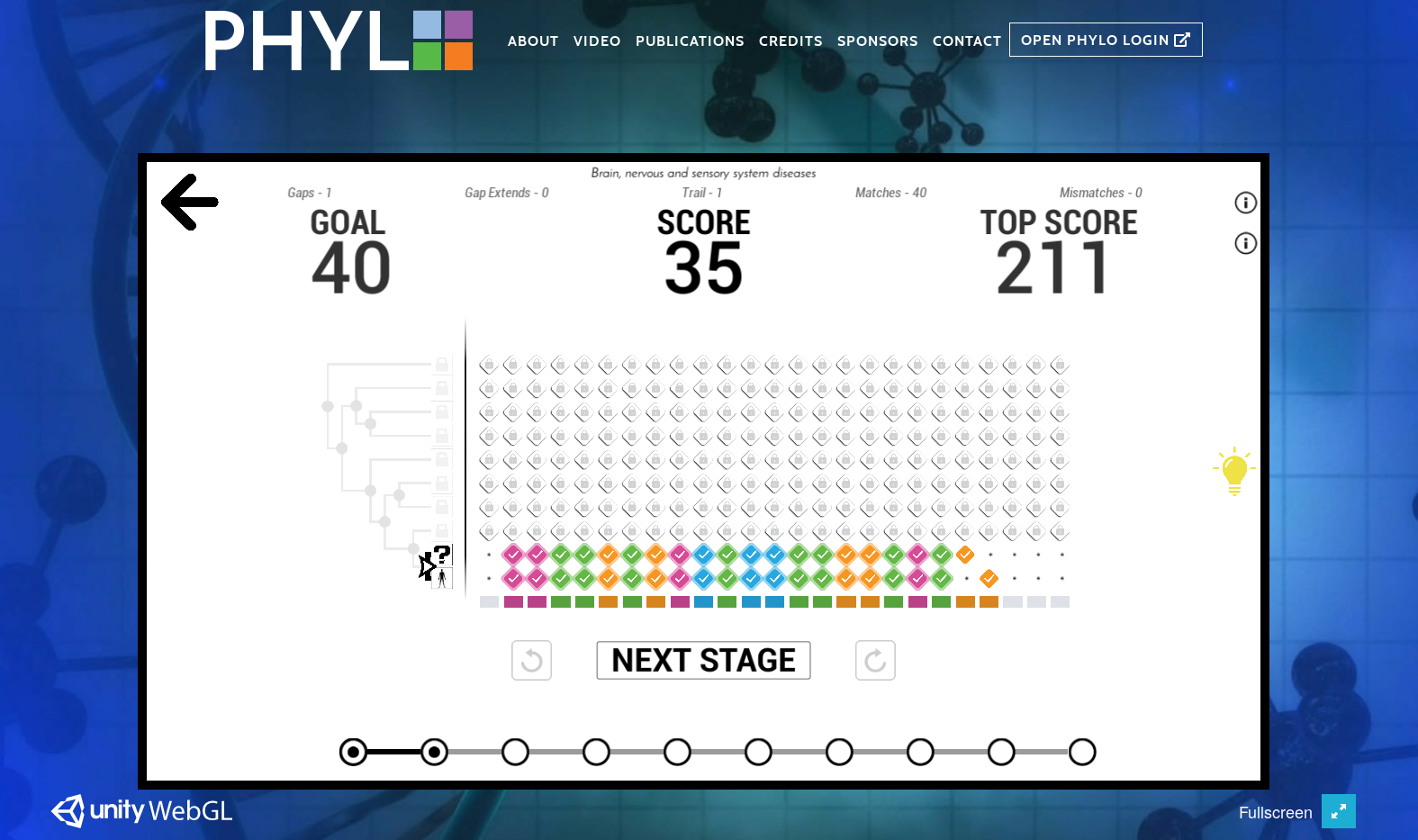
En date de 2017, plus de 400,000 joueurs distincts ont généré plus d’un million de solutions d’alignement (Singh et al., 2017). L’amélioration des alignements est de 36.7% lorsque l’agrégation est effectuée sur la base d’un arbre phylogénétique connu, et 66.3% si l’arbre phylogénétique est inconnu. Il est à mentionner que Phylo a été mis à contribution pour améliorer les alignements des souches du virus Ebola lors de l’épidémie de 2015 (Singh et al., 2017).
Avec la baisse des coûts de séquençage, le nombre de mutations rapportées pour les gènes a explosé. Or cette information n’est pas toujours répertoriée dans une base de données; le plus souvent elle est dissimulée dans la littérature (Burger et al., 2012). De plus, le traitement automatisé des résumés d’article avec des technique de NLP (Natural Language Processing) permet d’extraire les références aux mutations et aux gènes, mais pas de déterminer efficacement l’association entre eux. L’équipe de Burger et al. a voulu investiguer la possibilité de soumettre ces tâches d’association gène‑mutation à des non‑experts sur le marché de micro‑tâches Amazon Mechanical Turk (AMT). Pour chaque résumé d’article, un produit croisé des gènes et des mutations extraites du texte a été établit, puis chaque paire de gène‑mutation a été soumise en tant que HIT (Human Intelligent Task) sur AMT, et ce sous la forme d’une simple question demandant si la mutation est associée au gène ou non (Figure 4). Le contrôle de qualité est effectué par redondance et agrégation, mais également par l’insertion d’articles dont les associations sont connues.
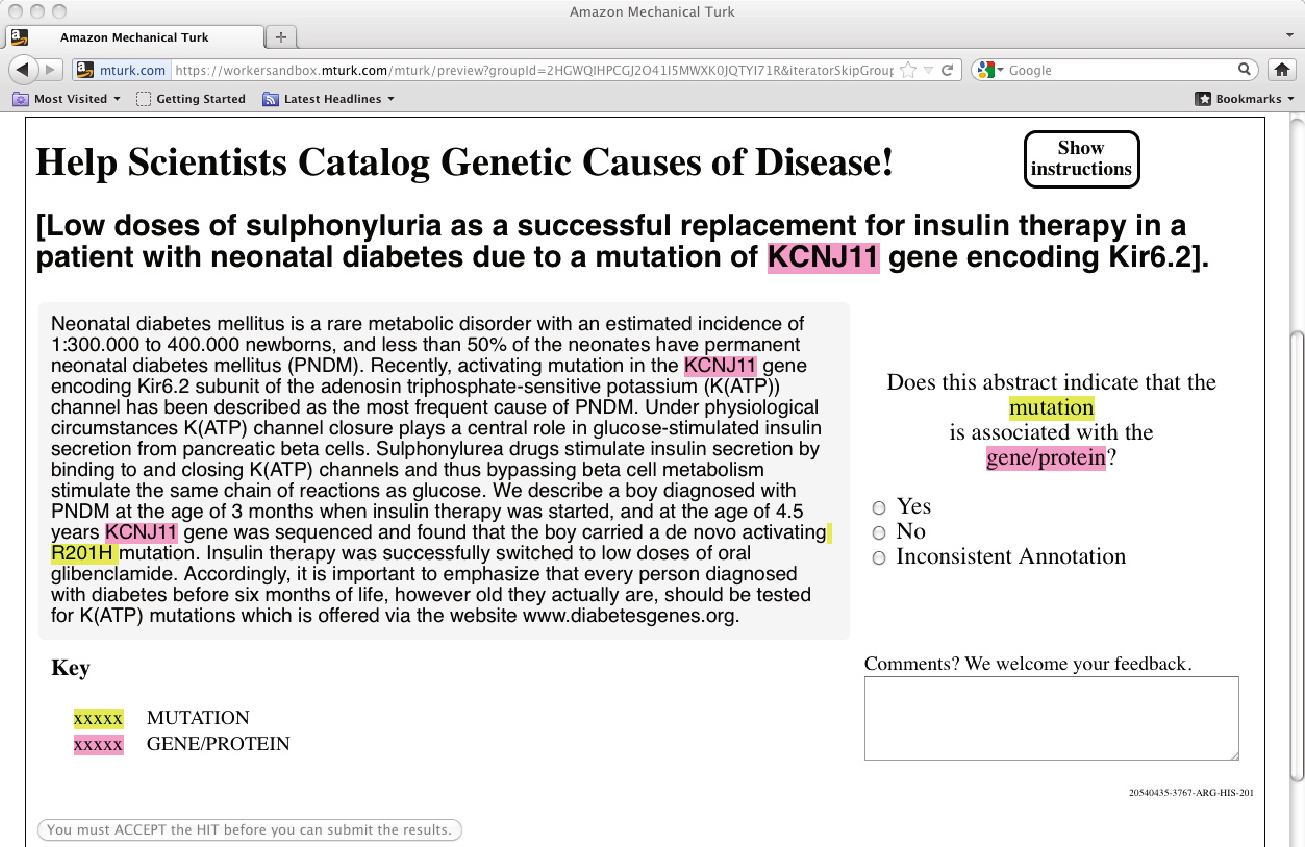
Un corpus de 250 résumés d’article a été sélectionné, produisant avec les contrôles 1,733 HITs soumis sur AMT à un taux de 7¢ US pour chaque HIT. Une contrainte de redondance de 5 travailleurs pour chaque HIT a été imposée, pour un total de 8,665 jugements. Ceux‑ci ont été complétés en moins de 36 heures par 23 répondants, et ce pour un coût d’opération de $670 US, incluant les frais d’Amazon. Les résultats, lorsque comparés à ceux des experts, montre une exactitude de 85% dans les associations entre mutation et gène, une performance qui est sujette à amélioration (Burger et al., 2012).
Un groupe de recherche a mobilisé une communauté de cliniciens de la ville de Houston, Texas, pour la collecte d’information concernant les liens entre problèmes médicaux et médicaments prescrits. Les bases de données existantes sont souvent peut maintenues, incomplètes, et n’utilise pas de terminologie commune (McCoy et al., 2012). De plus, l’extraction de ces informations des dossiers médicaux ou de la littérature à l’aide de techniques comme le data mining donne des résultats limités, souvent biaisés, ne répertoriant que les associations les plus courantes. La solution envisagée fût d’insérer dans le processus de prescription électronique de médicaments la saisie obligatoire d’une association avec un ou plusieurs problèmes cliniques (Figure 5), à l’instar de l’association obligatoire d’un mot à une ou plusieurs images avec ReCAPTCHA lors d’un accès sécurisé.
Les cliniciens ont manuellement associé 231,223 médicaments à des problèmes cliniques, produisant 41,203 liens distincts entre problème et médicament. En utilisant un seuil de pertinence de 95%, le nombre de paires a été restreint à 11,166. Une revue par des experts de la base de connaissance ainsi assemblée a déterminé une sensibilité de 65.8% et une spécificité de 97.9%, menant à la conclusion que cette méthode de crowdsourcing est efficace et peut coûteuse pour générer des bases de connaissance (McCoy et al., 2012).
La collecte de séquences métagénomiques pour l’étude de l’environnement représente une source inépuisable d’information. Or dans la plupart des cas les séquences n’ont pour annotations que le nom de l’auteur et la provenance géographique (Hingamp et al., 2008). L’Université d'Aix‑Marseille a choisi d’intégrer l’annotation de métagénome à son curriculum de bioinformatique, joignant le travail scientifique concret à l’apprentissage. La source de données choisie pour les séquences est le Global Ocean Sampling (GOS). Chaque étudiant se voit assigner la tâche d’annoter trois séquences, revues itérativement par un professeur. Un environnement spécifique — Annotathon (Figure 6) — a été développé pour la gestion et la revue des annotations. Les annotations de qualité sont au final publiées sur le site http://biologie.univ-mrs.fr/Metagenes/, accessible au public.
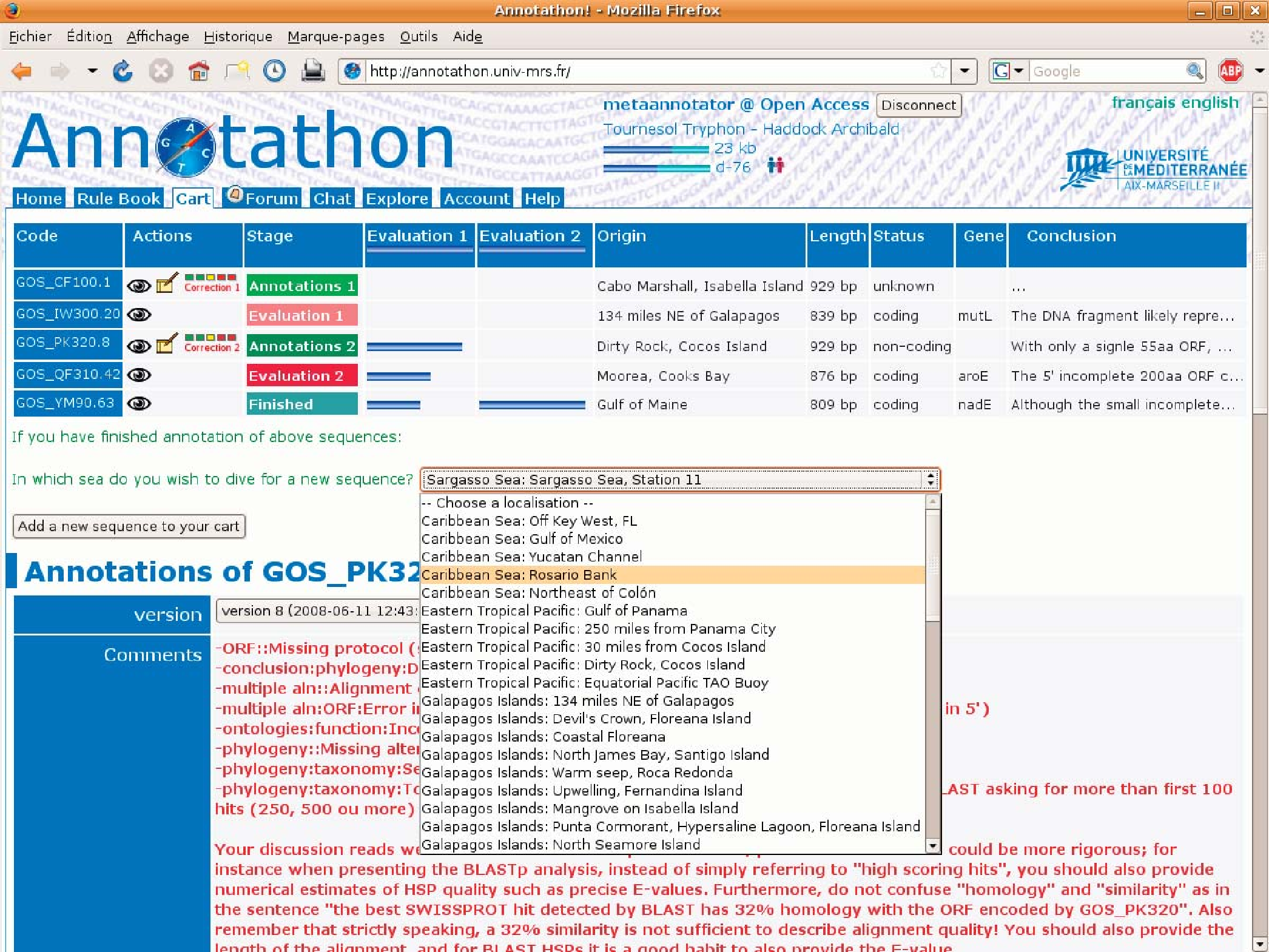
Après trois ans, 515 étudiants avaient pris part à l’analyse de 2.3Mb d’ADN microbien provenant des océans, et ce pour un total cumulatif de 9,500 heures d’annotation. Lorsque l’on compare les proportions d’assignations taxonomiques avec celles publiées dans la littérature sur GOS, on n’y retrouve pas de différences significatives. Malgré une différence entre les ontologies utilisées par GOS et Annotathon, il y a concordance des assignations fonctionnelles pour les quatre processus biologiques les plus abondants, soit transport, énergie, métabolisme de l’ADN et synthèse des protéines. L’expérience montre que l’intégration entre éducation et travail scientifique est positive pour l’apprentissage tout en produisant un résultat équivalent à celui des experts (Hingamp et al., 2008).
Le repliement d’une protéine est déterminé par sa structure d’acides aminés. Par contre, rechercher la conformation possédant un minimum d’énergie libre est une tâche computationnelle difficile étant donné le nombre de conformations possibles. Le jeu Foldit (https://fold.it/) propose d’associer les capacités de résolution de problèmes visuels et de développement de stratégies de joueurs non‑experts à une plateforme d’outils de manipulation de structures moléculaires nommée Rosetta (Cooper et al., 2010). Le joueur doit tenter d’obtenir le meilleur score, évalué à partir d’une fonction objective, par une combinaison de manipulations directes et l’application de petits algorithmes (Figure 7). Le jeu encourage la compétition, mais également la collaboration par l’échange de stratégies et d’algorithmes. Il s’agit d’un jeu qui demande un certain investissement; au départ, le joueur doit passer un grand nombre de niveaux d’entraînement, chaque niveau introduisant de nouvelles fonctionnalités ou possibilités.
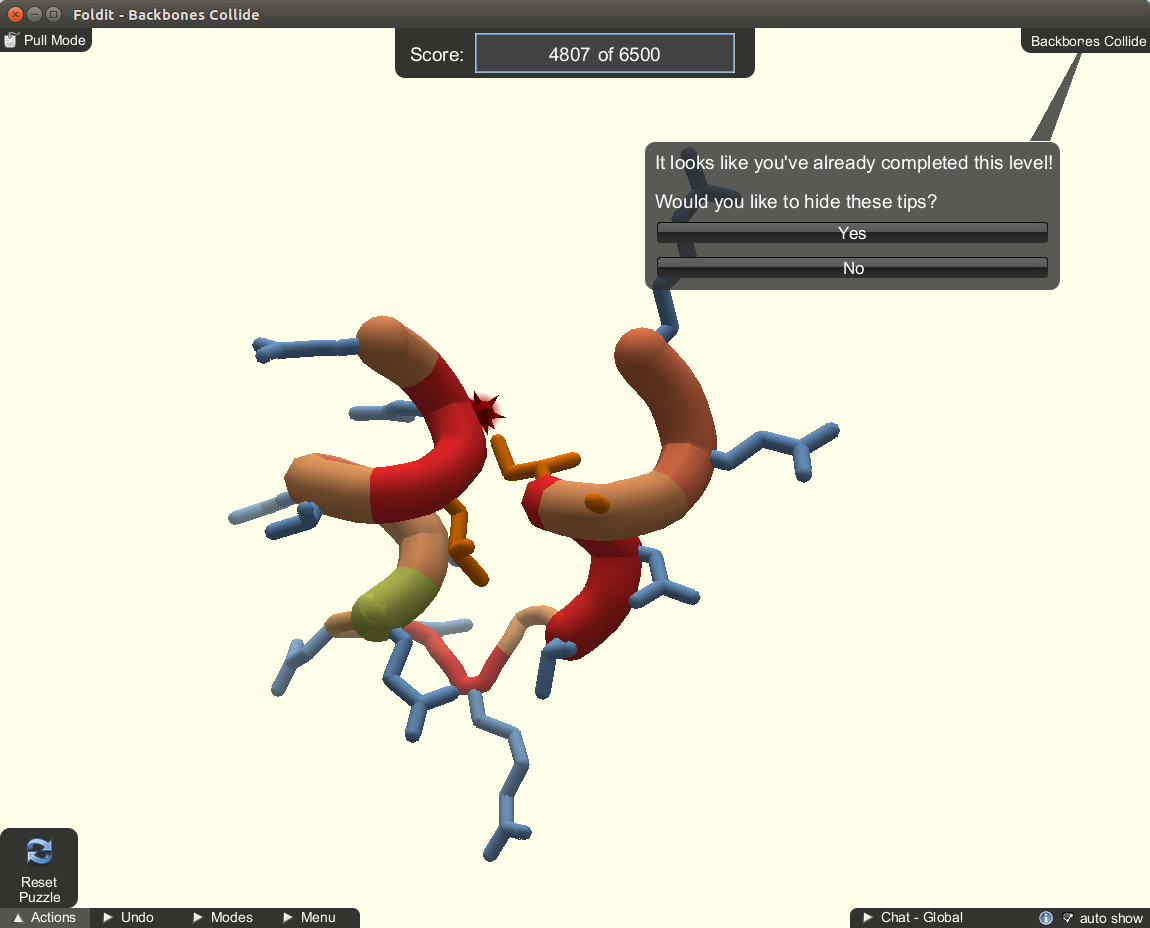
Foldit ne publie pas de statistiques explicites quant au nombre d’utilisateurs ou de problèmes résolus. Par contre, plusieurs succès ont été rapportés tels que la résolution de la structure d’un rétrovirus et l’optimisation d’enzymes à des buts physico‑chimiques spécifiques (Good et Su, 2013).
Les anticorps du système immunitaire ne sont pas le produit de gènes singuliers, mais le résultats d’une recombinaison continuelle de sections de gènes. De plus, les mutations sont courantes — insertions, délétions et substitutions. Il en résulte qu’un nombre relativement petit de segments de gènes peut produire un nombre énorme de molécules différentes, soit ~1030 (Lakhani et al., 2013). Le processus d’annotation consistant à déterminer avec précision quels segments ont contribué à une séquence devient un défi computationnelle avec un grand nombre séquences et les méthodes actuellement disponibles tels que BLAST et IMGT/V-QUEST. Le problème de trouver un algorithme plus performant a donc été soumis à un concours d’innovation, reformulé dans des termes non‑spécifiques au domaine de l’immunogénomique. Un jeu de données a également été assemblé pour la validation des résultats et mis à la disposition des compétiteurs. Enfin, une mesure de performance basée sur le temps d’exécution mais aussi sur l’exactitude des annotations a été conçue.
Le concours a été affiché sur la plateforme TopCoder.com, une communauté de plus d’un million de développeurs de logiciel qui compétitionnent régulièrement pour résoudre des défis de programmation. D’une durée de deux semaines, le concours offrait $6,000 US en prix, et recruta 733 participants dont 122 qui ont effectivement soumis du code de programmation. Parmi les soumissions, 30 ont excédé la référence de performance du US National Institute of Health MegaBLAST. La solution retenue a permis d’atteindre un plus haut niveau d’exactitude avec une vitesse de traitement 1,000 fois supérieure (Lakhani et al., 2013).
Le fait de déléguer le travail d’experts scientifiques à des non‑experts est aux fondements du crowdsourcing. Or s’agit-il d’un déplacement du siège de la connaissance vers les masses, ou simplement d’un déplacement de la force de travail? Les systèmes éducationnels ne font que remplacer des problèmes fictifs par des cas concrets dans le but reproduire la connaissance. Dans le cas des jeux et des concours d’innovation, on peut y voir un apport réel de créativité et d’imagination. Le volontariat est un choix. Mais l’utilisation des marchés de micro‑tâches est loin de faire l’unanimité. Par exemple, si l’on considère le nombre significatif d’heures passées par les travailleurs sur AMT en fonction de la rémunération, le salaire est dans certain cas inférieur à $2 US de l’heure (Good et Su, 2013). Ceci soulève des questions relevant de l’éthique : délègue-t-on le travail pour faire avancer la connaissance, ou simplement pour diminuer les coûts? Même avec les concours d’innovation il faut rappeler que seul les gagnants reçoivent une compensation; beaucoup d’autres aurons investi des quantités d’effort et de temps sans indemnité.
Finalement, même si le crowsourcing est considéré présentement comme une solution à certains problèmes, a‑t‑il un futur? Les limites computationnelles peuvent possiblement être solutionnée par d’autres systèmes apparentés, tel le grid computing. Quant aux capacités des technologies d’Intelligence Artificiel en regard des habiletés humaines, ils ne cessent de progresser. En fait, certaines données collectées par les méthodes de crowdsourcing pourrait potentiellement servir à l’entraînement de systèmes automatiques.
Cette étude a présenté une vue globale de l’application du crowdsourcing aux problèmes de bioinformatique. Les différents systèmes utilisés ont été décrits et un exemple concret pour chacun a été présenté. Les impacts sociaux et les alternatives existantes et futures ont été discutés. Il est à souligner que la présente investigation du sujet ne constitue en rien une étude exhaustive du crowdsourcing en bioinformatique, mais à la lumière des exemples mentionnés on peut conclure que le crowdsoursing appliqué à la bioinformatique semble être une solution viable pour certains types de problèmes.